DNA修復タンパク質が一本鎖DNAに特異結合する機構を解明 -高度好熱菌のDNA修復タンパク質「RecJ」を結晶化、X線結晶構造解析に成功-(プレスリリース)
- 公開日
- 2010年03月17日
- BL26B2(理研 構造ゲノムII)
2010年3月17日
独立行政法人理化学研究所
本研究成果のポイント
• RecJは一本鎖DNAを包み込むようなO型の構造を形成することが判明
• オリゴヌクレオチド/オリゴ糖結合フォールド構造が、一本鎖DNA親和性を生む
• DNA損傷が原因の疾病の解明や治療に新たな期待
|
独立行政法人理化学研究所(野依良治理事長)は、85℃という高温で生育し、進化の起源に近いと考えられる高度好熱菌サーマス・サーモフィラスHB8株※1を用いて、生命現象の根幹であるDNA修復機構※2の中で、一本鎖DNAを分解するタンパク質RecJの立体構造を明らかにし、一本鎖DNAに対する高い親和性の要因がRecJの構造によることを原子レベルで解明しました。これは、理研放射光科学総合研究センター(石川哲也センター長)放射光システム生物学研究グループの若松泰介連携研究員、倉光成紀グループディレクターらが「高度好熱菌丸ごと一匹プロジェクト※3」で行った研究成果です。 (論文) |
1.背 景
研究グループは、タンパク質をはじめとする生体分子の立体構造と生理・反応機能に基づき、1つの細胞すべての生命現象を、システム全体として理解しようと研究を展開しています。1999年には「高度好熱菌丸ごと一匹プロジェクト」を立ち上げ、モデル生物として、85℃という極限環境でも生育できる高度好熱菌サーマス・サーモフィラス HB8株を選びました。サーマス・サーモフィラスHB8株は、好熱菌の中でも(1)遺伝子数が約2,200個と少ない(ヒトは約23,000個、大腸菌は約4,500個)(2)厳しい環境で生きているためタンパク質が安定で、立体構造や機能を調べるのに都合がよい(3)遺伝子を操作する方法が確立されている、など多くの特徴を持っており、モデル生物に適していることが大きな理由です。
生命の遺伝情報はDNAに書き込まれており、これが複製エラーや紫外線などの外的要因で損傷し、書き換えられることは、進化の原動力となる一方で、細胞死や老化、がん化にもつながります。このため、生物はこの損傷を修復するためのさまざまなDNA修復機構を備えています。これらDNA修復を担うタンパク質は、細菌とヒトで異なりますが、その機構はほぼ共通しています。
研究グループはこれまで、この高度好熱菌を利用したDNA修復機構の解明に取り組んできました。高度好熱菌のDNA修復機構では、一本鎖DNA分解タンパク質がDNAのエラー箇所を排除するなど、非常に重要な役割を果たしていることが知られています。その中の1つに、細菌から多くの生物に共通して存在し、一本鎖DNAを分解する「RecJ」というタンパク質があり、DNA修復機構のうちミスマッチ修復機構※5(図1)や、塩基除去修復機構、相同組み換え修復機構で働くと考えられています。このRecJについては、1980年代中頃から主に大腸菌を用いた遺伝学的研究、生化学的研究は進んでいましたが、タンパク質の安定性が低く、結晶化が可能な良質なタンパク質を得ることが難しいため、立体構造は分かっていませんでした。このため、なぜ一本鎖DNAに対して高い親和性を持つ(結合する)のかも不明でした。
そこで、研究グループは、タンパク質が安定で結晶化効率も高いサーマス・サーモフィラスHB8株を活用し、RecJの一本鎖DNAに対する高い親和性の要因について、X線結晶構造解析を用いて明らかにすることを目指しました。
2.研究手法
良質な結晶を得るためには、タンパク質の安定性だけでなく、高純度の精製が可能であることが最低条件となります。研究グループは、精製時間の短縮や精製途中のタンパク質溶液への保存剤の添加など、精製方法の改良を進めることで、精製度を高めることに成功し、さらに、幅広い結晶化条件のスクリーニングと最適化により、解析可能な結晶を得るに至りました。この結晶について、高分解能でイメージングを行うため、SPring-8の理研 構造ゲノム II ビームラインBL26B2を用いてX線結晶構造解析を行いました。
3.研究成果
SPring-8の高輝度な放射光によって、2.15Å(オングストローム)※6という高分解能のX線解析データを得ることができました。これらのデータを解析し、RecJ単体の構造だけではなく、触媒活性発現(DNAを分解すること)に必須なマグネシウムイオンやマンガンイオンとの、複合体構造を明らかにすることに成功しました。この解析から、RecJが一本鎖DNAを分解するのに適した、特別な構造をしていることが明らかになりました。
(1) RecJは4つのドメインからなるO型の構造をしている
X線結晶構造解析により、RecJは2つのドメインからなる触媒活性発現に必須なコアドメイン(ドメインIとII)と、そのほかに2つのドメイン(ドメインIIIとIV)を持ち、これら4つのドメインが環状につながってO型の構造を形成していることが分かりました(図2)。また、ドメインIとIIの間にある触媒活性部位の大きさは約11Åで、一本鎖DNA(直径約10Å)を分解するのに適した構造をしていることを明らかにしました。
(2) RecJは典型的なDNA結合構造を含んでいる
RecJは一本鎖DNA分解タンパク質ですが、そのドメインIIIには、DNA結合タンパク質に典型的な構造として知られる、オリゴヌクレオチド/オリゴ糖結合フォールド※7を持っていました(図3)。さらに、この部分を機能しないように変化させたRecJとの働きを比較するため、ゲルシフトアッセイ※8と呼ばれる生化学的解析を行いました。解析の結果、ドメインIIIが実際に一本鎖DNAと結合することが分かり、この領域が一本鎖DNAとの高い親和性に寄与することを証明しました。一本鎖DNA分解タンパク質に、オリゴヌクレオチド/オリゴ糖結合フォールド構造が含まれるということは、これまでに予想すらされていませんでした。DNA解析でRecJのアミノ酸配列はすでに知られていましたが、この構造が含まれることは推測できておらず、今回の構造解析で初めて明らかとなりました。
(3) 一本鎖DNAとの新たな結合モデルを提案
これまでの知見と今回の研究成果を基に、RecJと一本鎖DNAとの結合モデルを構築しました(図4)。RecJは、コアドメインとオリゴヌクレオチド/オリゴ糖結合フォールドのDNA結合構造が一本鎖DNAを包み込むようなO型の構造をしているため、一本鎖DNAと結合する時は、これまでに知られていた一本鎖DNA分解タンパク質には見られない、ユニークな構造であることが予測されます。
4.今後の期待
RecJの立体構造を原子レベルで明らかにしたことで、一本鎖 DNA分解タンパク質が、なぜDNAに対して高い親和性を示すのか、その理由はDNAを包み込むようなO型構造や、オリゴヌクレオチド/オリゴ糖結合フォールド構造の存在によることが明らかとなりました。多くの生物には、RecJ とよく似たアミノ酸配列を持つ一本鎖DNA分解タンパク質が存在しますが、ほとんどがその構造や機能は不明です。今回の成果は、今後、それらの一本鎖DNA分解タンパク質の機能や構造を考える上で非常に役立つ知見となり、DNA修復機構の解明に大きく寄与することになります。DNA修復機構の仕組みは、細菌からヒトまで共通であると考えられることから、このような高度好熱菌を用いたDNA修復機構の解明は、将来的には、がんやDNA損傷を原因とする疾病の解明、治療につながることが期待されます。
《参考資料》
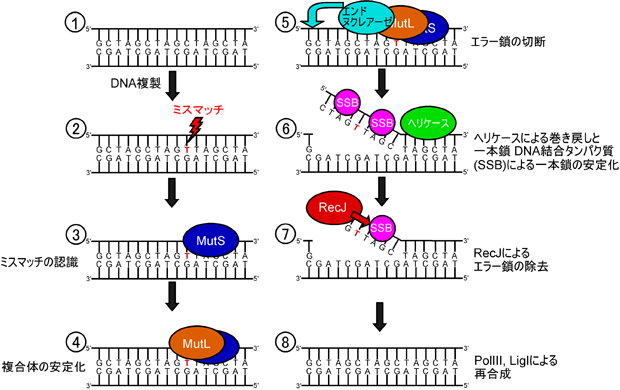
DNAが複製する時に( )、何らかの原因でミスマッチエラー(本来はTでは無くCが結合)が起こると(
)、何らかの原因でミスマッチエラー(本来はTでは無くCが結合)が起こると( )、タンパク質MutSがミスマッチを見つけDNAに結合する(
)、タンパク質MutSがミスマッチを見つけDNAに結合する( )。そこにタンパク質MutLが結合し(
)。そこにタンパク質MutLが結合し( )、さらにDNA分解タンパク質エンドヌクレアーゼがエラーを含む一本鎖DNAを切断(
)、さらにDNA分解タンパク質エンドヌクレアーゼがエラーを含む一本鎖DNAを切断( )、タンパク質ヘリケースがその一本鎖DNAをはがすとともに、一本鎖DNA結合タンパク質(SSB)が一本鎖DNAと結合して再び二重らせんDNAにならないようにする(
)、タンパク質ヘリケースがその一本鎖DNAをはがすとともに、一本鎖DNA結合タンパク質(SSB)が一本鎖DNAと結合して再び二重らせんDNAにならないようにする( )。SSBがタンパク質RecJに結合してエラー鎖を分解し(
)。SSBがタンパク質RecJに結合してエラー鎖を分解し( )、タンパク質Pol IIIやタンパク質Lig Iによって二重らせんDNAが再合成される(
)、タンパク質Pol IIIやタンパク質Lig Iによって二重らせんDNAが再合成される( )。
)。
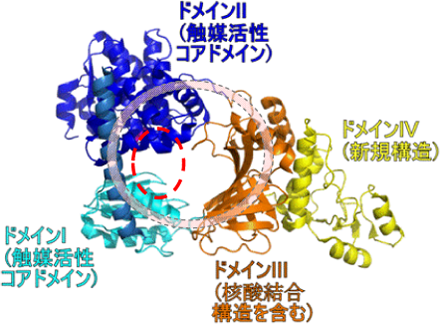
ドメインIとIIからなる触媒活性コアドメインと、DNA結合タンパク質に典型的なオリゴヌクレオチド/オリゴ糖結合フォールドを含むドメインIII、新規構造でその役割はいまだ不明であるドメインIVの4つのドメインが環状につながることによってO型の構造を形成している。触媒活性部位を赤の点線で示した。
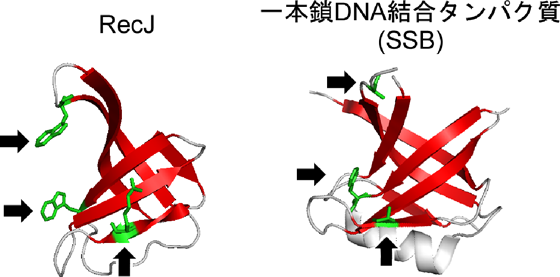
オリゴヌクレオチド/オリゴ糖結合フォールド
RecJのドメインIIIの中には、一本鎖DNA結合タンパク質(SSB)と同様に、一本鎖DNAと相互作用するアミノ酸残基が存在する(緑色部分)。一本鎖DNA結合に関与する部位(赤色のリボン)の構造が両者でよく似ている。
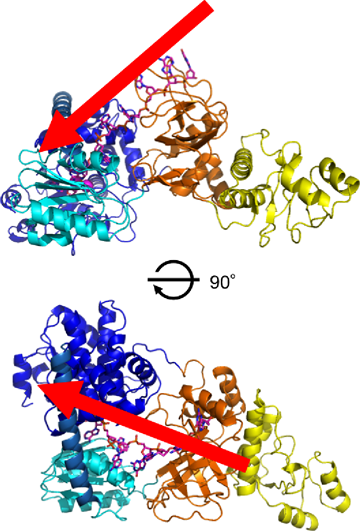
ピンク色がモデル一本鎖DNAを表す。一本鎖DNAは赤矢印の方向(ドメインIII中のDNA結合構造から、ドメインIおよびIIのコアドメイン方向へ)で進入し、触媒活性部位で分解されると予想できる。下図は、上図を前後方向に90度回転させたもの。
《用語解説》
※1 高度好熱菌サーマス・サーモフィラスHB8株
静岡県伊豆半島にある峰温泉から発見された、85℃という極限環境で生育できる細菌。熱水中で生きている細菌(好熱菌)は全生物の共通祖先に近い位置にあり、原始生命の基本的特徴が凝縮されていると考えられている。好熱菌一匹に起こる生命現象を理解することは、ヒトを含めたあらゆる生物の基本を解明し、「生命とは何か」という本質的な疑問を問い直すことにつながるとも考えられている。
※2 DNA修復機構
DNAは絶えず複製エラーや活性酸素種などの内的要因と、紫外線や電離放射線の照射などの外的要因により損傷を受けている。しかし、生物はこれら損傷を元通りに修復するさまざまなDNA修復機構を備えており、この機構には多くのDNA修復タンパク質がかかわっている。今回立体構造を明らかにしたRecJは、ミスマッチ修復(※5参照)、塩基除去修復、相同組み換え修復で働くと考えられている。
※3 高度好熱菌丸ごと一匹プロジェクト
高度好熱菌サーマス・サーモフィラスHB8株を、地球上のあらゆる生物の代表(モデル生物)とし、DNA、タンパク質、糖質、脂質、そのほかの低分子の構造と機能に基づいて、1つの細胞におけるあらゆる生命現象をシステムとして理解する学問基盤の構築を目指すプロジェクト。このプロジェクトは以下の4段階で進行すると想定しており、SPring-8においてはイメージングに関連した研究を行う。
第1段階:タンパク質など細胞を構成する分子の、1つの細胞全体の立体構造解析
第2段階:タンパク質など細胞を構成する分子の、1つの細胞全体の機能解析
第3段階:細胞内のそれぞれのシステム(複数分子のネットワーク関係)の解析
第4段階:細胞全体のシミュレーション
※4 大型放射光施設SPring-8(スプリングエイト)
理研が所有する、兵庫県の播磨科学公園都市にある世界最高の大型放射光施設。SPring-8 の名前はSuper Photon ring-8 GeVに由来する。放射光(シンクロトロン放射)とは、電子を光とほぼ等しい速度まで加速し、電磁石によって進行方向を曲げたときに発生する、細く強力な電磁波 のことである。SPring-8では、遠赤外から可視光線、軟X線を経て硬X線に至る幅広い波長域で放射光を得ることができるため、原子核の研究からナノテクノロジー、バイオテクノロジー、産業利用や科学捜査まで幅広い研究が行われている。SPring-8は日本の先端科学・技術を支える高度先端科学施設として、日本国内外の大学・研究所・企業からの年間1万4,000人以上の研究者によって利用されている。
※5 ミスマッチ修復機構
DNA損傷の修復方法の1つ。DNAはアデニン(A)、グアニン(G)、シトシン(C)、チミン(T)の4種類の塩基がそれぞれ、A-T、C-Gと結合して二重らせんを形成している。DNAが複製される時、二重らせんは解かれ、それぞれの一本鎖DNAに新たな塩基が結合して、2本の新しい二重らせんDNAを形成する。ミスマッチ修復機構は、DNAが複製する時に生じた誤った塩基の組合せ(ミスマッチ)を見つけ出し、除去する修復機構。
※6 Å(オングストローム)
1オングストロームは1×10-10メートル(=0.1ナノメートル)。X線結晶構造解析においては、立体構造の分解能を示す単位として用いられる。数字が小さいほど、より高分解能であることを示す。
※7 オリゴヌクレオチド/オリゴ糖結合フォールド
オリゴヌクレオチドは、約20塩基対或いはそれ以下の長さの短いDNAまたはRNAの配列。オリゴ糖は、約20あるいはそれ以下の多糖が結び付いたもの。オリゴヌクレオチド/オリゴ糖結合フォールドは、DNAと結合する典型的な構造として知られ、そのフォールドに含まれる数種類の特徴的なアミノ酸残基が、DNAとの結合に関与している。
※8 ゲルシフトアッセイ
DNAをはじめとする核酸とタンパク質の結合を調べる実験方法のこと。リンの放射性同位体で核酸を標識し、タンパク質と混合した後、電気泳動により放射線を検出することで核酸の移動度を測定する。遊離した核酸は速く流れるため下部にバンドが現れるが、タンパク質が結合した核酸は遅く流れるため上部にバンドが現れる。これによりタンパク質と核酸の結合量を測定する。
《報道担当・問い合わせ先》 播磨研究推進部 企画課 (報道担当) (SPring-8に関すること) |
- 現在の記事
- DNA修復タンパク質が一本鎖DNAに特異結合する機構を解明 -高度好熱菌のDNA修復タンパク質「RecJ」を結晶化、X線結晶構造解析に成功-(プレスリリース)