クライオ電子顕微鏡で酵素タンパク質の動的構造を可視化 -従来の酵素反応理論を立体構造から見直す成果-(プレスリリース)
- 公開日
- 2024年05月29日
- BL38B1(理研 構造生物学 I)
- クライオ電子顕微鏡(EM01CT)
2024年5月29日
理化学研究所
|
理化学研究所(理研)放射光科学研究センター生命系放射光利用システム開発チームの中迫雅由客員主管研究員、若林大貴研修生、大出真央客員研究員(研究当時)の研究チームは、クライオ電子顕微鏡[1]観察によって動作中に現れる複数の酵素タンパク質[2]の立体構造を可視化することに成功しました。 論文情報 |
反応中のグルタミン酸脱水素酵素で検出された4状態のクライオ電子顕微鏡マップ(右3状態が酵素-補酵素-基質複合体)
背景
酵素タンパク質は、あらゆる生物体内に存在し、生命活動に不可欠な化学反応とそれに伴う分子輸送などを担っています。その立体構造は、これまで、X線結晶構造解析[4]によって解明されてきました。そこで得られる原子レベルの立体構造は、結晶化に必要な特殊な溶媒環境下のものであり、また、結晶配列を維持する相互作用によって運動性に制限を受けています。そのため、反応時に生じる酵素タンパク質の大規模な構造変化を可視化できる可能性が低くなります。
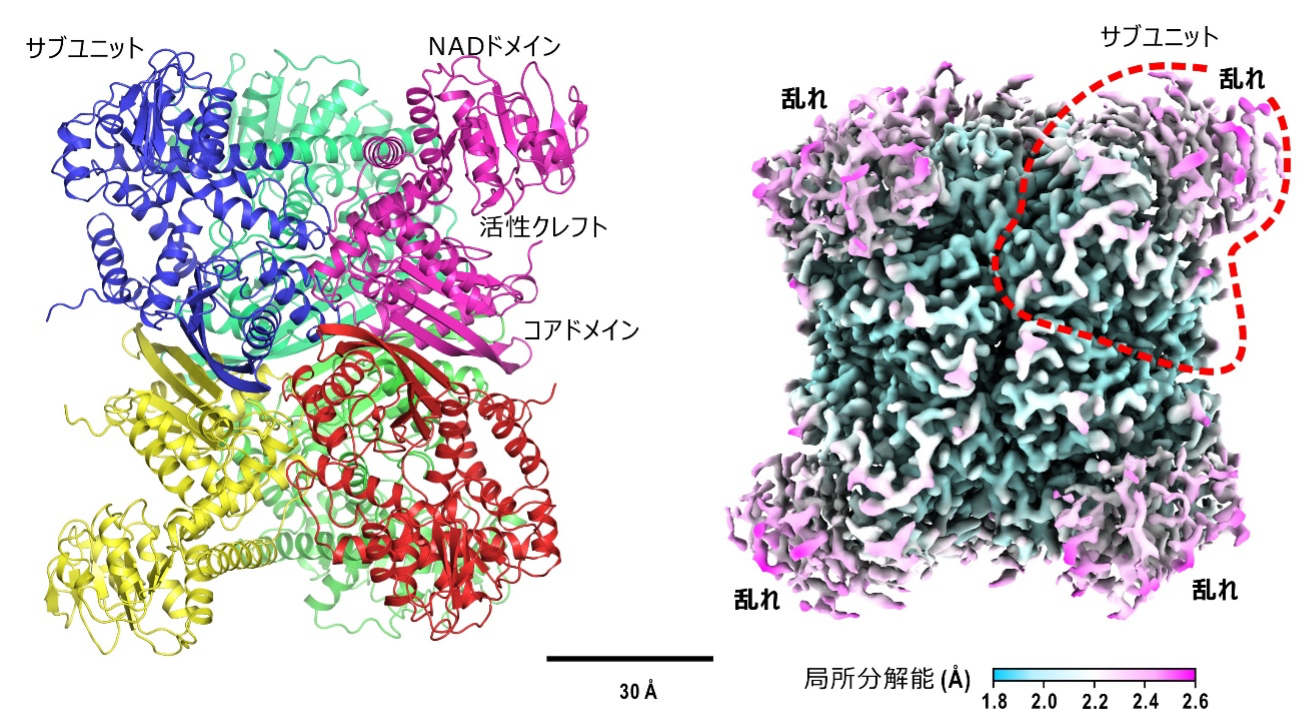
酵素タンパク質の一つである本成果で対象としたグルタミン酸脱水素酵素は、419個のアミノ酸で構成されたサブユニットが6個会合して機能単位となります。サブユニットは、化学反応を補助する補酵素[5]を結合するためのNADドメインと、六量体を形成するコアドメインに分かれており、これら二つのドメインの間に基質[6]を結合して化学反応が行われる活性クレフトが形成されています(図1左)。この酵素が何も結合していない状態の立体構造については、これまでにX線結晶構造解析、X線小角散乱法[7]や分子動力学計算[8]によって調べられており、NADドメインがコアドメインに、近づいたり離れたりする1ナノメートル(nm、1nmは10億分の1メートル)程度のちょうつがい運動をし、その制御には、活性クレフトの最も奥まったところで発生する水分子の乱雑な脱着が関与していました注1)。しかし、この酵素がどのような構造変化をして化学反応に至るのかは不明でした。
図1 グルタミン酸脱水素酵素六量体の結晶中および水溶液中の立体構造
(左)グルタミン酸脱水素酵素の結晶構造モデル。(右)クライオ電子顕微鏡で得られた水溶液中の静電ポテンシャル像。NADドメイン部分が複数の構造にあることを示す乱れが認められた。
研究手法と成果
クライオ電子顕微鏡は、水溶液中のタンパク質の立体構造を調べるのに適しています。また、水溶液中でタンパク質がさまざまな立体構造をとる場合には、画像分類法[9]を適用することで、代表的な立体構造の電子顕微鏡画像を抽出分類して、各立体構造を可視化できます。今回、研究チームは、グルタミン酸脱水素酵素の酵素反応中にどのような立体構造変化が生じるのかを調べるため、酵素、補酵素、基質の各溶液を混合して急速凍結し、そこに現れている立体構造を調べました。この観察には、 大型放射光施設「SPring-8」[10]サイトに設置されたクライオ電子顕微鏡を用い、また、凍結によって酵素の構造が変化しないこと確認するため、凍結前の溶液中の大まかな構造をBL38B1のX線小角散乱法で解析しました。
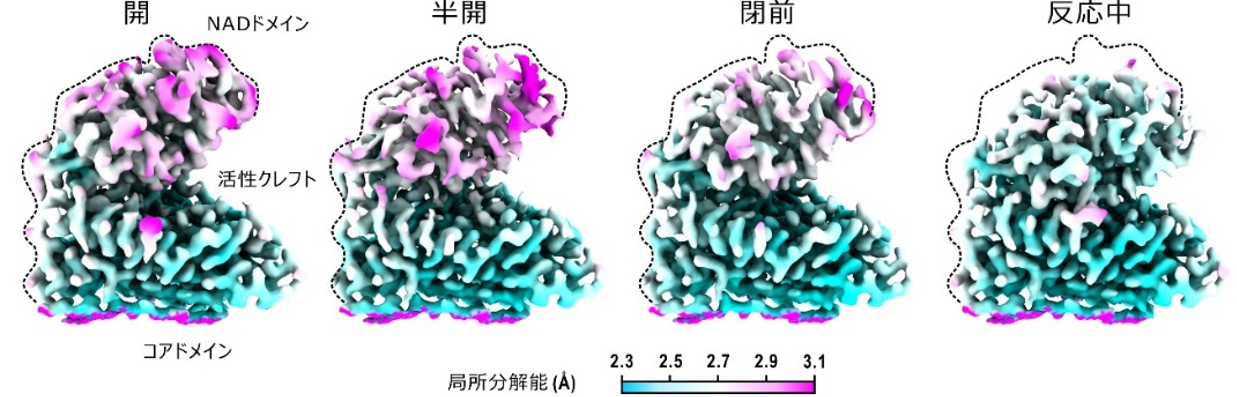
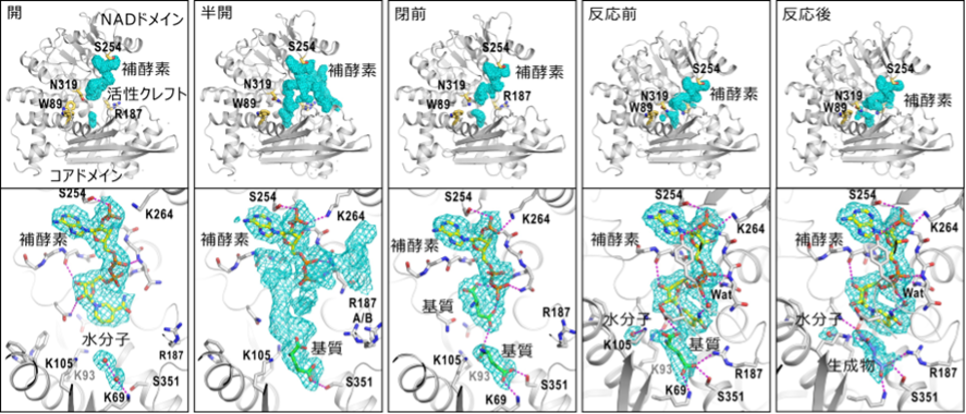
通常の電子顕微鏡単粒子構造解析法[9]によってグルタミン酸脱水素酵素六量体の立体構造を得たところ、NADドメインの静電ポテンシャルマップに乱れがあり、複数の立体構造が示唆されました(図1右)。そのため、単粒子構造解析法の一手法である画像分類法によって、NADドメインのコアドメインに対する位置をさらに詳しく解析したところ、反応初期の段階には補酵素分子が結合した四つの立体構造が現れました。それらは、活性クレフトが開いた状態(開)、基質が結合して活性クレフトが少し閉じた状態(半開)、基質が結合してさらに閉じた状態(閉前)、そして完全に閉じて化学反応が行われている状態でした(反応中)。特に、反応中状態では、反応前と反応後の構造を見分けることができました。これらの立体構造を基に、化学反応サイクルを立体構造から解析することが可能になりました(図2)。それによれば、(1)溶液混合後すぐに補酵素はNADドメインに結合し、その後に基質が結合する。しかし、補酵素末端領域が揺らぐため、基質が結合してもすぐにグルタミン酸脱水素酵素が、化学反応が行える状態にはならない(半開と閉前)、(2)補酵素末端領域が活性クレフトを閉じるNADドメインの運動を阻害しない状態で化学反応に適した状態が実現できる(反応中)、(3)反応が終わると活性クレフトが開いて生成物を放出し、水分子2個を結合して次の基質結合に備える(開)というものです。
図2 4状態における活性クレフト内の補酵素、基質、生成物と水分子の状態
上段にはサブユニットのどこに補酵素が結合しているのかを明示し、その拡大図を下段に示す。水色のメッシュは静電ポテンシャルマップで、タンパク質分子はリボンモデル、補酵素と基質/生成物は棒モデルで表示し、それらと相互作用しているアミノ酸残基と水素結合(紫破線)を表示してある。
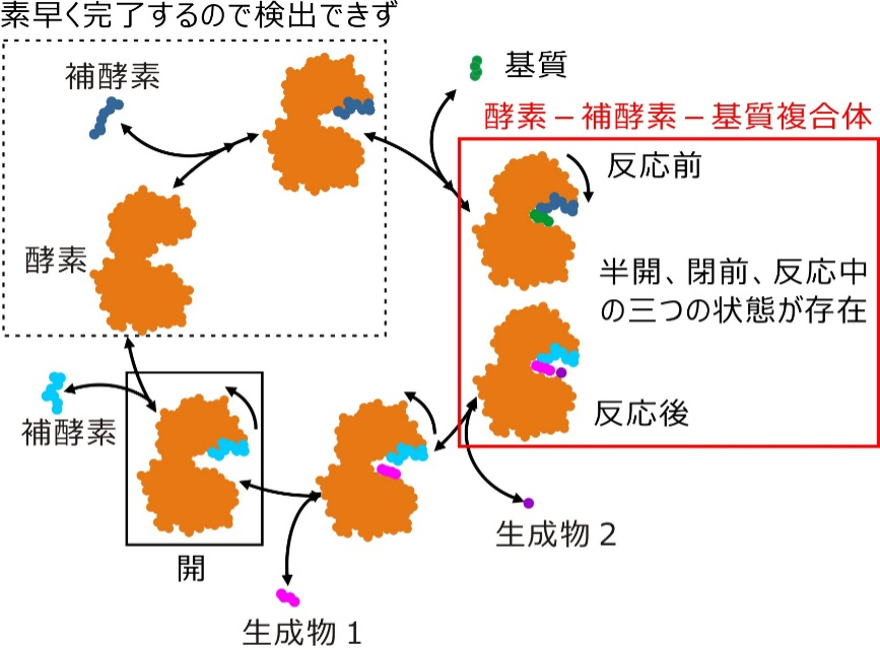
グルタミン酸脱水素酵素の反応に酵素反応速度論[11]を適用する場合、酵素-補酵素-基質複合体を一つの状態として取り扱いますが、今回の構造解析の結果、この一つの状態は、NADドメインと補酵素の運動によって三つの状態(半開、閉前、反応中)が混じったものであることが明らかとなりました(図3)。
図3 立体構造から見た酵素反応
今回の構造解析で可視化できた状態を実線枠、できなかった状態を点線枠で示した。酵素反応速度論では、反応前後の酵素-補酵素-基質複合体(赤枠)を単一構造と見なして解析し、それ以上の構造状態についてさらに詳細な解析はできない。今回の構造解析では、単一構造として取り扱われてきた状態が三つの構造の混合状態であることを明らかにした。
注1)2016年5月20日慶應義塾大学プレスリリース「タンパク質分子のナノスケール運動が水和水分子の脱吸着で制御されることを明らかに」
今後の期待
今回の研究チームの成果は、水溶液中でのタンパク質の運動や機能発現過程を可視化する上でクライオ電子顕微鏡が威力を発揮し得ることを明らかにしました。結晶環境ではないため、得られた立体構造を、そのまま大規模な分子動力学計算によってさらに詳しく解析することができます。また、このような方法を用いれば、薬剤が酵素タンパク質に作用する過程を可視化することが可能になるかもしれません。また、研究チームが開発してきたタンパク質の表面に吸着している水和水分子位置(タンパク質の水和構造[12])を予測できる人工知能(AI)[13]、注2)を用いれば、水分子とドメイン運動の関わりを解析でき、より完全な酵素反応の構造実態を記述することが可能になると期待できます。
注2)2023年2月22日プレスリリース「タンパク質の水和構造を予測する人工知能」
【用語解説】
[1] クライオ電子顕微鏡
タンパク質などの生体分子を水溶液中の生理的な環境に近い状態で、電子顕微鏡で観察するために開発された手法。試料を含む水溶液を急速凍結して、液体窒素温度(-196℃)付近まで冷却することで電子線の照射による損傷を低減し、生体分子やその複合体の構造解析を行うことができる。電子線の波長は可視光よりもはるかに短いため、理論上0.1nm程度の分解能が得られる。近年、急速に発展した手法で、この構造解析技術を開発した科学者は2017年のノーベル化学賞を受賞した。
[2] 酵素タンパク質
酵素タンパク質は、生命活動に不可欠な物質を変換する化学反応の触媒として機能する分子である。酵素タンパク質には、さまざまなものがあり、例えば、食物の消化から代謝まで、筋肉の収縮、遺伝子の転写や複製など、ありとあらゆる生命現象の素過程を担っている。
[3] グルタミン酸脱水素酵素
古細菌から真核生物まで広く分布する尿素合成に必須な酵素タンパク質で、基質グルタミン酸とα-ケトグルタル酸の相互変換反応を触媒する。この酵素が働くためには、触媒反応を補助するための補酵素が不可欠である。なお、本研究で使用したグルタミン酸脱水素酵素は、日本海溝の海底火山から採取された超高度好熱古細菌から単離されたものである。
[4] X線結晶構造解析
対象となる分子を結晶化し、結晶にX線を照射して得られるX線回折現象から、結晶内分子の3次元構造を可視化する実験手法。理研が所有する兵庫県の播磨科学公園都市にある世界最高性能の放射光を生み出す大型放射光施設「SPring-8」([10]参照)では、生体高分子のX線結晶構造解析が精力的に行われている。
[5] 補酵素
酵素タンパク質に可逆的に結合・解離する有機化合物で、酵素タンパク質にはない活性部位を有して触媒反応を助ける。ビタミンは典型的な補酵素である。
[6] 基質
酵素タンパク質に結合し、触媒反応によって他の化学物質に変換される分子。
[7] X線小角散乱法
X線を物質に照射して、散乱するX線のうち散乱角が小さいものを測定することで物質の構造情報を得る手法。タンパク質や核酸などの生体高分子、微粒子や液晶、合金の内部構造といった数ナノメートル(nm)での構造解析に用いる。例えば、超臨界流体のような系では、粗密な領域から成る不均一構造(密度ゆらぎ)や密度ゆらぎの相関距離といった構造パラメータにより、その中・長距離構造を定量的に評価できる。
[8] 分子動力学計算
原子や分子の動きを計算機上で解析するための方法。生体高分子の場合には、水分子集団に内包された生体高分子が時間経過に伴う構造変化をどのように行うのかを、古典力学における運動方程式を数値的に解くことによって決定する。粒子間に働く力は、力場パラメーターで決められている。タンパク質のような巨大分子への分子動力学計算の適用に貢献した計算科学者には、2013年にノーベル化学賞が授与された。理研が所有するスーパーコンピュータ「富岳」では、新型コロナウイルスのスパイクタンパク質をはじめとして、創薬分野に関わる分子動力学研究が精力的に行われている。
[9] 画像分類法、単粒子構造解析法
単粒子構造解析法は、ほぼ同一の構造を持つ粒子をさまざまな角度からランダムに撮影した多数の2次元画像から、信号/雑音比(S/N比)を向上させ、その3次元構造を再構成する方法。透過型電子顕微鏡における生体分子の構造解析において発展してきた。単粒子構造解析法では、通常、同じ構造をさまざまな角度から見たと仮定して解析を行うが、同じ構造ではなく、異なった構造が混在している場合、図1右のような乱れた像を与える。そのため、複数の構造が存在する場合には、単粒子構造解析で求めた像の中で、複数の構造をとり得る部分に注目して、同じ構造になっている像を分類し、各構造ごとに立体構造解析を行う。これを画像分類法と呼ぶ。
[10] 大型放射光施設「SPring-8」
兵庫県播磨科学公園都市にある世界最高性能の放射光を生み出す理研の実験施設。SPring-8(スプリングエイト)の名前はSuper Photon ring-8 GeVに由来する。放射光(シンクロトロン放射)とは、電子を光とほぼ等しい速度まで加速し、電磁石によって進行方向を曲げたときに発生する細くて強力な電磁波のこと。SPring-8では、遠赤外線から可視光線、軟X線を経て硬X線に至る幅広い波長域で放射光が得られるため、原子核の研究からナノテクノロジー、バイオテクノロジー、産業利用や科学捜査まで幅広い研究が行われている。
[11] 酵素反応速度論
酵素タンパク質によって触媒される化学反応の生成物量を測定し、単位時間当たりどのぐらいの反応生成物が生じているのか(反応速度)を解析する理論。酵素反応の機構や薬物による酵素反応阻害について解析できる。グルタミン酸脱水素酵素の触媒反応では、酵素-補酵素-基質複合状態が単一であるとして解析できるが、実際には、今回明らかになったように、複数の状態が混在する。
[12] タンパク質の水和構造
タンパク質分子は、水溶液や脂質の中で特定の立体構造を形成する。また、その機能発現には、一定量以上の水分子がタンパク質に吸着している必要があることが知られてきた。低温X線結晶構造解析が開発されたことを契機に、タンパク質表面の水分子の吸着状態が原子レベルで可視化されるようになり、この表面やタンパク質内部の水分子の配列様態をタンパク質の水和構造と呼ぶ。水和構造は、タンパク質の構造安定化、分子認識、機能発現において不可欠な役割を担っていることが明らかにされている。
[13] 人工知能(AI)
ヒトの知能を研究する情報工学分野や、そこで創生される計算機上のシステム。創生されるシステムは、事例を基に訓練され、新たに与えられる問題の解決に向けた認識、推論、言語運用および創造などの知的行動を人間に代わって行う。情報工学分野で創生されてきた人工知能は、基礎および応用科学分野だけではなく、現在、日常的にも応用されるようになっている。AIはArtificial Intelligenceの略。
研究支援
本研究は、日本学術振興会(JSPS)科学研究費助成事業基盤研究(B)「クライオ電顕画像から蛋白質の動的構造を描写するための新規計算科学手法の確立と応用(研究代表者:中迫雅由)」の助成を受けて実施されました。
|
発表者・機関窓口 |
- 現在の記事
- クライオ電子顕微鏡で酵素タンパク質の動的構造を可視化 -従来の酵素反応理論を立体構造から見直す成果-(プレスリリース)
 ml.riken.jp
ml.riken.jp